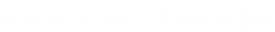iTRAQ标记定量
同位素标记相对和绝对定量(iTRAQ) 蛋白质组学技术
Isobaric Tags for Relative and Absolute Quantization (iTRAQ) proteome technology
同位素标记相对和绝对定量iTRAQ(Isobaric tags for Relative and Absolute Quantitation)技术是由美国应用生物系统公司(Applied Biosystems Incorporation,ABI)开发的一种多肽体外标记技术。该技术采用了4种( iTRAQ® reagents – 4plex)或8种( iTRAQ® reagents – 8plex )同位素标签,通过标记肽段(Peptide)特异氨基酸位点,然后进行串联质谱分析,可灵活比较最多8种不同样本中蛋白质的相对含量和绝对含量。

技术原理 技术特点 技术流程 FAQ 产品信息 成功案例
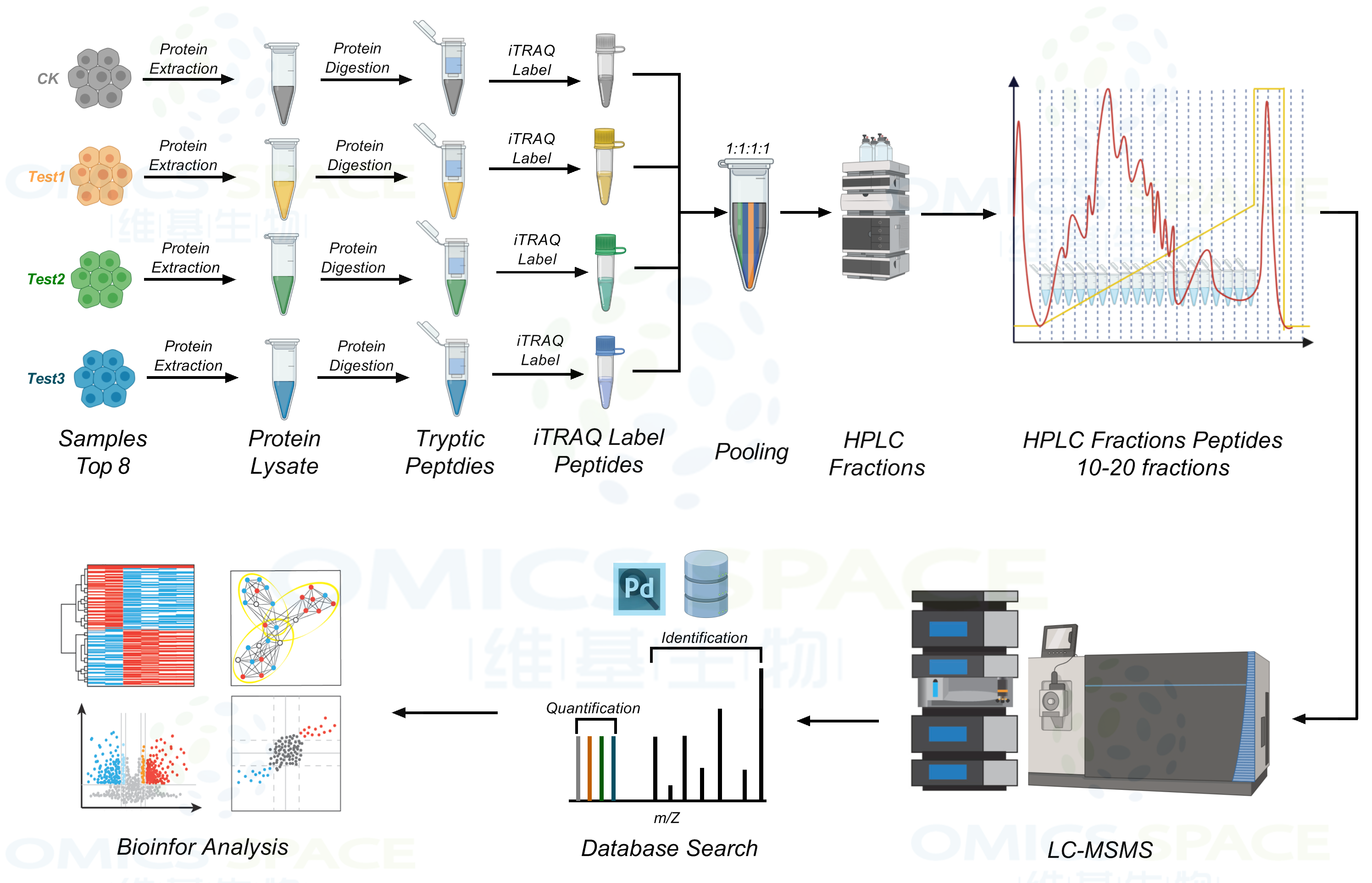
iTRAQ实验中,经过蛋白质提取获得组织细胞中的全蛋白,全蛋白在胰酶作用下被酶解(Trypsin Digestion)成肽段,iTRAQ试剂对所获得的全部肽段进行标记。iTRAQ试剂主要由3部分组成:报告基团(Report group)、平衡基团(Balance group)和肽反应基团(Amine-specific reactive group)。以4标试剂为例:报告基团相对分子质量分别为114、115、116和117;平衡基团相对分子质量分别为31、30、29和28,肽反应基团将iTRAQ试剂与肽段上赖氨酸(Lysine,K)及N端氨基酸残基相连,在其上4种报告基团通过平衡基团与反应基团相连,这样就形成了4种相对分子质量均为145的等量异位标签。
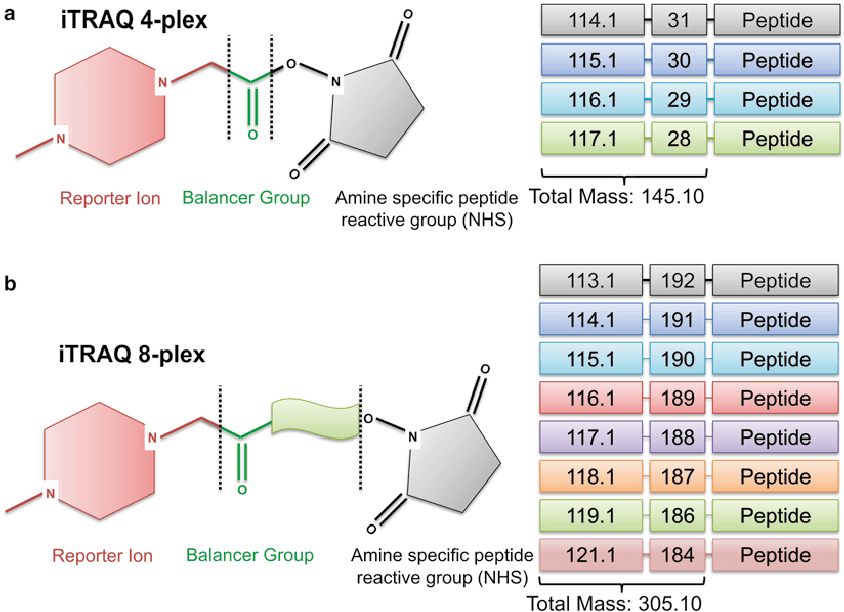
Figure reference:Aggarwal, Suruchi, and Amit Kumar Yadav. "Dissecting the iTRAQ data analysis." Statistical Analysis in Proteomics. Humana Press, New York, NY, 2016. 277-291.
iTRAQ试剂在标记肽段样本后,报告基团和平衡基团的平衡分子量都为145,因此改变任一iTRAQ试剂,不同同位素标记的同一多肽在第一级质谱检测中分子量都完全相同。而在串联质谱(MSMS)中,同重元素标记肽段的报告基团、平衡基团和肽反应基团之间的连接键断裂,平衡基团中性丢失,同时不同同位素标签的同一肽段产生不同的质荷比(114-117)的峰。因此,根据波峰的高度和面积,可获得样品间相同肽段的定量信息,再经过软件处理得到肽段和蛋白质的定量信息。
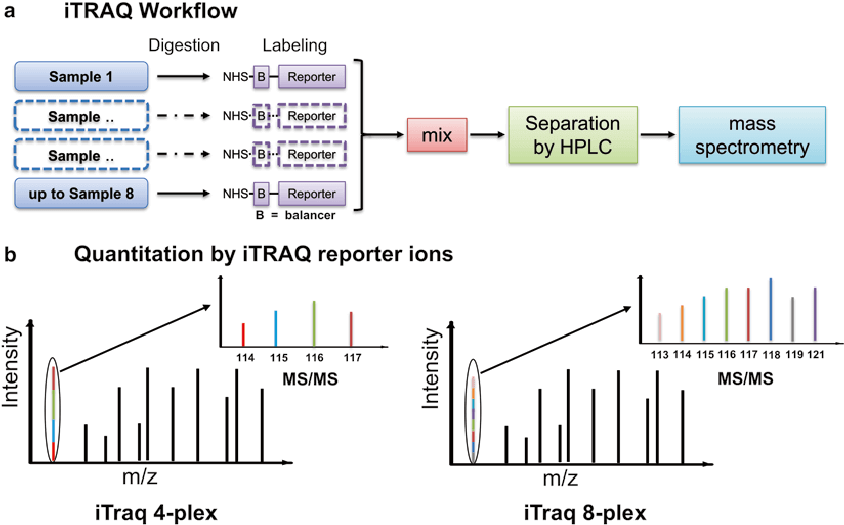
Figure reference:Aggarwal, Suruchi, and Amit Kumar Yadav. "Dissecting the iTRAQ data analysis." Statistical Analysis in Proteomics. Humana Press, New York, NY, 2016. 277-291.
参考文献:
1. Wu, Wells W., et al. "Comparative study of three proteomic quantitative methods, DIGE, cICAT, and iTRAQ, using 2D gel-or LC− MALDI TOF/TOF." Journal of proteome research 5.3 (2006): 651-658.
2. Evans, Caroline, et al. "An insight into iTRAQ: where do we stand now?." Analytical and bioanalytical chemistry 404.4 (2012): 1011-1027.
3. Köcher, Thomas, et al. "High precision quantitative proteomics using iTRAQ on an LTQ Orbitrap: a new mass spectrometric method combining the benefits of all." Journal of proteome research 8.10 (2009): 4743-4752.
4. Gan, Chee Sian, et al. "Technical, experimental, and biological variations in isobaric tags for relative and absolute quantitation (iTRAQ)." Journal of proteome research 6.2 (2007): 821-827.
优点:
1.通量高,一次iTRAQ实验可同时获得几千蛋白质的定量和定量信息。
2.一次实验可同时完成对多个样本的测试。
3.提高了二级质谱的碎片效率。
4.减少了整个实验周期以及样本之间的实验误差。
5.数据的重现性较高。
6.更有利于蛋白质互作关系以及表达模式的分析。
缺点:
1.实验成本较高。
2.对样本中含盐量的耐受程度较低。
3.需要特殊的分析软件。
4.样品酶解效率与实验误差紧密相关。
1. 关于样品重复的问题?
最优的实验设计是每组样品设置至少三个生物学重复,比如有A和B两组,最优是提供A1、A2、A3、B1、B2、B3共6个样品;其次是技术重复,只有A和B两个样本,没有生物学重复,推荐做A和B 各3次平行的质谱技术重复。最后是否做生物学重复或者技术重复,还可以有一些其他考虑:如果实验本身在整个研究中处于后期的验证,或者是一个前期的开始,可以考虑1次重复;类似血清或者分泌液的样本也可以做1次重复。无论最后的重复如何设计,一个严谨的设计都需要后期对潜在蛋白质的验证,比如Realtime-PCR,Western-Blot,ELISA等。
2. iTRAQ实验一次可以做多少个样本?
理论上利用一个8标试剂盒,一次实验最多可以做8个样本。但如果在所有的实验里面设置内标的话,就可以把多个8标实验关联起来,这样就可以做超过8个样本的iTRAQ实验了。
3.通过iTRAQ找到的候选蛋白会与其他蛋白的变化趋势保持一致吗?
做为一个高通量的实验,iTRAQ最主要的目的是发现一些可能有意义的潜在蛋白,对表型的表现从蛋白的层面上去找一些可能的证据,是一个“发现”过程,需要后期通过其他的实验区再次验证。所以,在一个iTRAQ实验开始之前,我们不会就某个特殊的蛋白质的检出,以及显著性和变化倍数等做出保证。
4. 为什么要设计预备实验?
设置预备实验室将整个iTRAQ实验的风险降至最低。通过预备实验结果我们就可以大致判断最后整体实验的情况。比如有些菌类样本,即便在SDS-PAGE图,以及质谱的Basepeak图都是一样的,可以搜完库之后鉴定得到的蛋白质数据会相差比较大,出现这种情况就很有可能是该菌有其他杂菌的污染,如果没有预备实验,这样的情况可能会比较难排查。
1.产品描述:iTRAQ标记定量比较蛋白质组学-对客户提供的不同组别的样本进行蛋白质组学分析,鉴定获得所有样本的肽段/蛋白信息,定量筛选不同组别样本之间显著性差异表达的蛋白质,并对显著性差异蛋白质进行生物信息学分析。
2.样品要求:
2.1. 常规人、动物组织(比如肝脏、肾脏、脑组织等):≥100mg;
2.2. 软体动物:≥100mg;
2.3. 动物坚韧组织(软骨、毛发等):≥500mg;
2.4. 植物叶片等鲜嫩组织:湿重≥1g;
2.5. 植物富含杂质或蛋白含量低的样本,如植物根,根茎、木质部、韧皮部组织等:干重≥2g;
2.6. 植物花粉:≥100mg;
2.7. 藻类组织:≥1g;
2.8. 细胞样本数目须达到:105(建议使用本公司的裂解液进行裂解之后再进行寄送);
2.9. 微生物菌类:≥50µL纯菌体;
2.10. 体液类(唾液、羊水、脑脊液等)1 mL以上,不能溶血;血清50 μl以上;尿液10 mL以上。
2.11. 其他特殊样本如有疑问请联系我们。
3.质谱信息:Orbitrap Fusion(Thermo Scientific),Orbitrap Elite(Thermo Scientific)
4.测试周期:25个工作日
5.报告信息:测试报告、肽段/蛋白质鉴定列表(电子版)、显著性分析列表(电子版)、生物信息学报告、原始质谱测试文件
6:生信分析:
6.1. 质谱数据质量分析
6.2. 显著性差异统计分析
6.3. GeneOntology注释和富集分析
6.4. KEGG注释和富集分析
6.5. 蛋白质互做网络PPI分析
Alterations of brain quantitative proteomics profiling revealed the molecular mechanisms of diosgenin against cerebral ischemia reperfusion effects.
Zhang X, Wang X, Khurm M, et al.
Journal of Proteome Research, 2020.
Tandem mass tag-based quantitative proteomic analysis of lycorine treatment in highly pathogenic avian influenza H5N1 virus infection.
Yang L, Zhang J H, Zhang X L, et al.
PeerJ, 2019, 7: e7697.
Quantitative proteomics analysis reveals resistance differences of banana cultivar ‘Brazilian’ to Fusarium oxysporum f. sp. cubense races 1 and 4
Honghong Dong, Yunfeng Li, Huiyun Fan, Dengbo Zhou, Huaping Lia
Journal of Proteomics 203 (2019) 103376
Comparative analysis of proteomic and metabolomic profiles of different species of Paris.
Liu F, Meng Y, He K, Song F, Cheng J, Wang H, Huang Z, Luo Z, Yan X.
Journal of proteomics. 2019 Mar 16.
Proteomic analysis of the rice (Oryza officinalis) provides clues on molecular tagging of proteins for brown planthopper resistance
Xiaoyun Zhang, Fuyou Yin, Suqin Xiao, Chunmiao Jiang, Tengqiong Yu, Ling Chen, Xue Ke, Qiaofang Zhong, Zaiquan Cheng and Weijiao Li
BMC Plant Biology 201919:30
Quantitative Proteomics of Potato Leaves Infected with Phytophthora infestans Provides Insights into Coordinated and Altered Protein Expression during Early and Late Disease Stages
CX, JG,YZ,DZ,QC et al.and YS
Int. J. Mol. Sci. 2019, 20(1)
iTRAQ-Based Differential Proteomic Analysis Reveals the Pathways Associated with Tigecycline Resistance in Acinetobacter baumannii.
Yang, N., Liu, Y., He, P., Ke, R., Zhao, Y., Feng, Y., ... & Wu, X.
Cellular Physiology and Biochemistry, 2018, 51(3): 1327-1339.
Proteomic Profiling Analysis of Male Infertility in Spodoptera Litura Larvae Challenged with Azadirachtin and its Potential‐Regulated Pathways in the Following Stages.
Sun, R., Cui, G., Chen, Y., Shu, B., Zhong, G., & Yi, X.
Proteomics, 2018, 18(19): 1800192.
Cyanobacteria blooms induce embryonic heart failure in an endangered fish species.
Zi, J., Pan, X., MacIsaac, H. J., Yang, J., Xu, R., Chen, S., & Chang, X.
Aquatic Toxicology, 2018, 194: 78-85.
Differentiation Model Establishment and Differentiation-Related Protein Screening in Primary Cultured Human Sebocytes.
Zhang M F, Cai X L, Jing K P, et al.
BioMed research international, 2018, 2018.
Immune-related proteins detected through iTRAQ-based proteomics analysis of intestines from Apostichopus japonicus in response to tussah immunoreactive substances.
Mi R, Sun Y, Li J, et al.
Fish & shellfish immunology, 2018, 74: 436-443.
Chemical proteomics reveal CD147 as a functional target of pseudolaric acid B in human cancer cells.
Zhou Y, Di Z, Li X, et al.
Chemical Communications, 2017, 53(62): 8671-8674.
iTRAQ Proteomic Analysis Reveals That Metabolic Pathways Involving Energy Metabolism Are Affected by Tea Tree Oil in Botrytis cinerea.
Xu J, Shao X, Wei Y, et al.
Frontiers in microbiology, 2017, 8: 1989.
Proteomic analysis of differentially expressed proteins in the two developmental stages of Ichthyophthirius multifiliis
Yao JY, Xu Y, Yuan XM, Yin WL, Yang GL, Lin LY, Pan XY, Wang CF, Shen JY
Parasitol Res. 2017 Feb;116(2):637-646. doi: 10.1007/s00436-016-5328-3