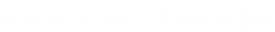资讯中心
MCP2017-三角褐指藻脂肪酸代谢途径的乙酰化蛋白质组学分析
发布日期:2018-04-27 10:39 浏览: 3,494 views次

赖氨酸乙酰化是一种高度动态、可逆调控的翻译后修饰,广泛存在于几乎所有生物体内,在多种代谢途径中对蛋白质功能的调控起着重要作用。尽管在包括细菌、酵母、植物和人类在内的几种生物中,乙酰化蛋白的蛋白质组分析已被报道,但对光合作用微藻中的乙酰化蛋白组了解甚少。微藻被视为极富前景的生物柴油新原料。硅藻是海洋浮游植物的主要类群,也是海洋生态系统中的主要初级生产者。硅藻代表藻种三角褐指藻(Phaeodactylum tricornutum)因其光合效率高、脂质含量高且基因组序列已公布,成为研究热点。在本文中,作者使用蛋白质组学方法分析了三角褐指藻中的赖氨酸乙酰基。总共鉴定了来自1220个乙酰化蛋白的2324个乙酰化位点,这是迄今为止植物中赖氨酸乙酰化组的最大数据集,且几乎所有参与脂肪酸合成的酶都被发现是赖氨酸乙酰化的。作者分析发现位点特异性赖氨酸乙酰化对酰基辅酶a合成酶活性的调节,并强调了赖氨酸乙酰化对三角褐指藻脂肪酸代谢的潜在调节作用。
1.乙酰化修饰结果概述
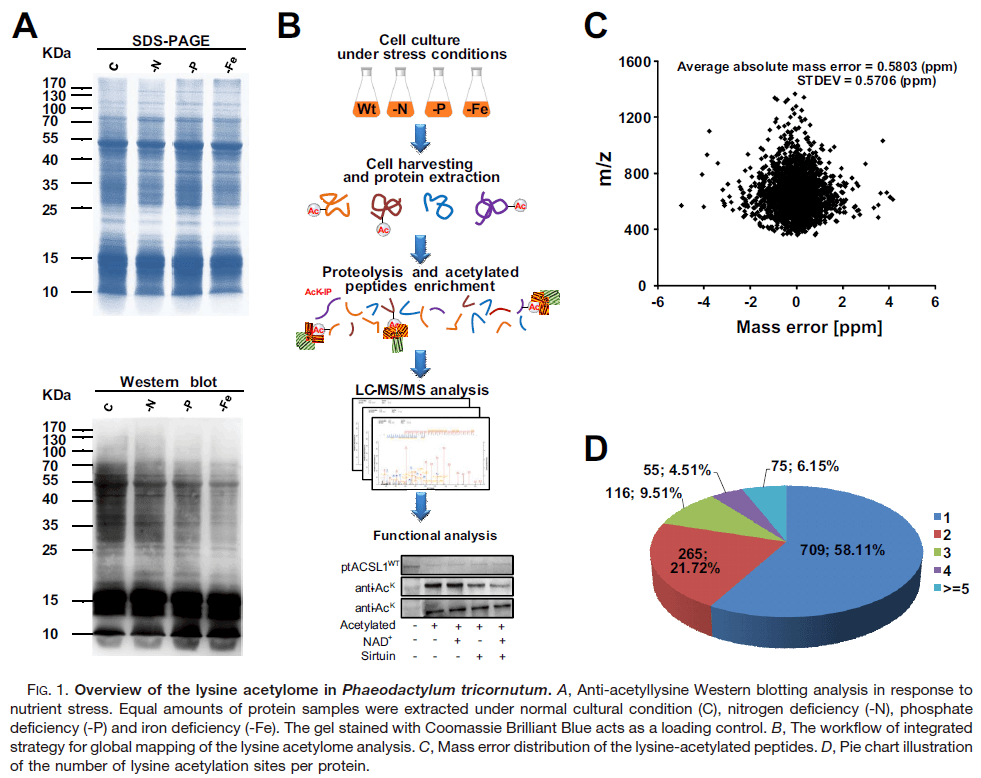
首先对三角褐指藻中的赖氨酸乙酰基进行了大规模的分析,分析了在不同生长条件下(正常培养条件下(C)、缺氮(-N)、缺磷(-P)和缺铁(-Fe)条件下)三角褐指藻全细胞裂解产物中乙酰化肽。实验之前通过WBt实验发现这4个处理乙酰化模式有明显的不同(图1A);之后采用2D-LC/MS/MS进行乙酰化修饰蛋白组学(图1B)共鉴定得到1220个乙酰化蛋白和2324个乙酰化位点。其中超过6.15%的蛋白质携带至少五个识别位点,包括两个长链酰基辅酶A合成酶(ptACSL1和ptACSL4)(图1D)。
2. 乙酰化蛋白功能分析
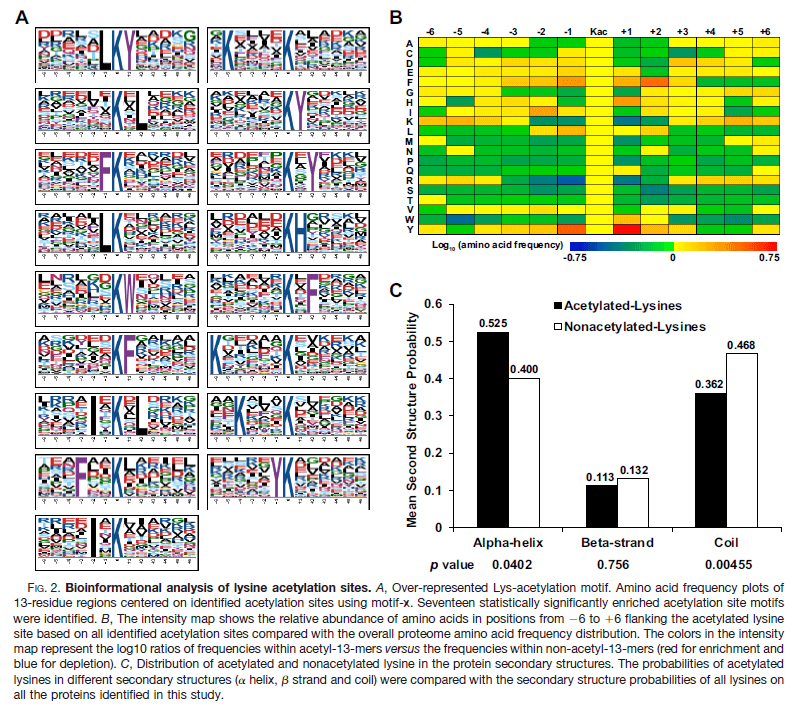
对鉴定得到乙酰化修饰位点通过motif-x将位点上游和下游的六个侧位氨基酸频率与该模型中所有赖氨酸残基的频率进行了比较,发现17个显著富集的乙酰化位点基序,占81.5%。其中三个基序,Y,H或F在朝向C末端的+1位置上的富集,其中Y在+1上伴随着L在-1位置或L在+2/I在-1位置首次在植物中被识别(图2A)。通过热图分析(图2B)发现乙酰化肽的残基偏好在-1和+1位置为Y,在-1/+1/+2位置为F,在-1/+2位置为L,这些都与motif-x分析的先前结果一致。这些数据显示了一些氨基酸残基对三角褐指藻乙酰化的偏好。对赖氨酸乙酰化与乙酰化蛋白局部二级结构的关系进行二级结构分析。发现乙酰化位点在α-螺旋中的分布约为52.5%,在β-片状中的分布约为11.3%,在螺旋中的分布约为36.2%(图2C)。这一发现表明,赖氨酸乙酰化可能更倾向于在光合有机体的α-螺旋结构。
3. 重要酶乙酰化修饰WB验证
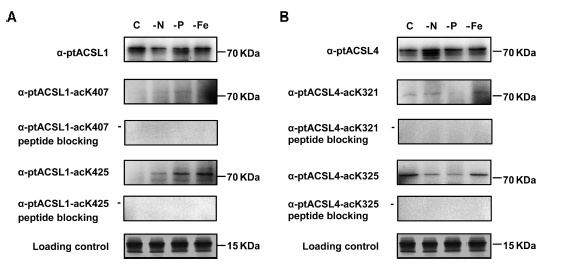
三角褐指藻通过II型脂肪酸合成途径合成脂肪酸。实验发现该途径7种酶发生乙酰化。包括关键第一限速酶ACCase,长链酰基辅酶a合成酶(LACS)等。之前研究表明LACS的ptACSL1和ptACSL4有促进外源脂肪酸摄取的功能。本实验发现ptACSL1和ptACSL4被大量的Lys乙酰化。WB确认ptACSL1和ptACSL4的赖氨酸乙酰化(K407、K425、K321和K325),同时发现赖氨酸乙酰化的ptACSL1和ptACSL4表现出不同于营养胁迫下序列特异性抗体识别的表达模式(图3)。
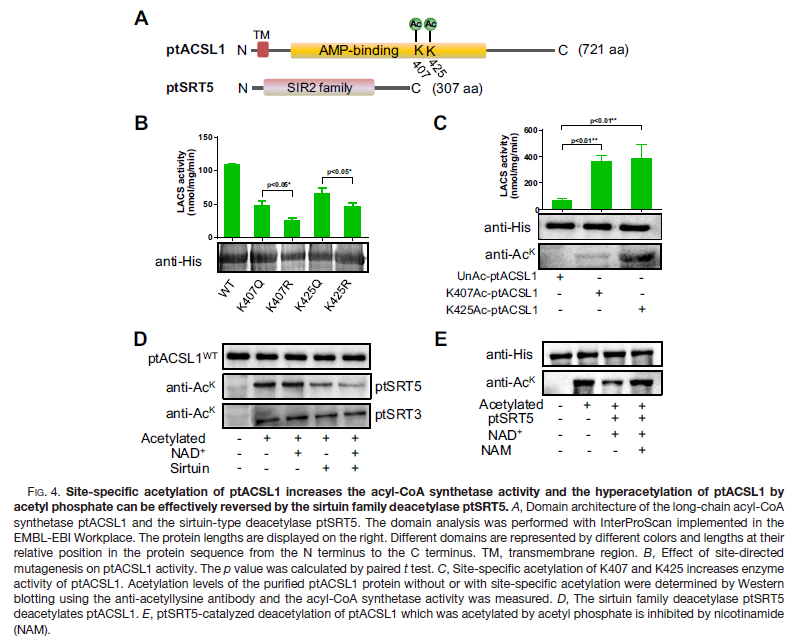
LACS定点突变的活性实验。将ptACSL1的位于AMP结合区的K407和K425分别突变为谷氨酰胺(K407Q,K425Q),谷氨酰胺(K407Q,K425Q)。酶活结果表明所有的定点突变都导致LACS活性下降(图4B)。通过大肠杆菌重组纯化K407-,K425-乙酰化,和未乙酰化的ptACSL1,酶活发现与未乙酰化的ptACSL1相比,K407和K425的乙酰化导致ptACSL1的LACS活性显著增加(图4C)。
Sirtuin是ptACSL1的一种常见的去乙酰化酶。纯化Sirtuin(ptSRT5)并于乙酰化的ptACSL1进行孵育,发现ptACSL1在NAD+存在下能有效地脱乙酰,而ptSRT3在同一反应体系中不能诱导ptACSL1脱乙酰(图4D)。如果添加烟酰胺(SIRT脱乙酰化酶家族的特异性抑制剂)可完全抑制ptSRT5对乙酰化ptACSL1的脱乙酰化活性。以上表明AcP乙酰化的ptACSL1可以被三角褐指藻中SIRT脱乙酰酶家族的sirtuin蛋白ptSRT5逆转。
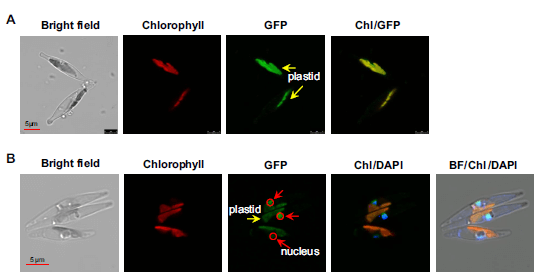
通过构建ptACSL1和ptSRT5全长开放阅读框的GFP融合体,发现ptACSL1和ptSRT5都能定位在叶绿体中,推测ptACSL1可能通过可逆的Lys乙酰化和ptSRT5在叶绿体中的去乙酰化来调节。